Crystal structure of pneumolysin from Streptococcus pneumoniae
Park, S.A., Lee, K.S.To be published.
Experimental Data Snapshot
Starting Model: experimental
View more details
wwPDB Validation 3D Report Full Report
Entity ID: 1 | |||||
|---|---|---|---|---|---|
| Molecule | Chains | Sequence Length | Organism | Details | Image |
| Pneumolysin | 484 | Streptococcus pneumoniae | Mutation(s): 0 Gene Names: ply | 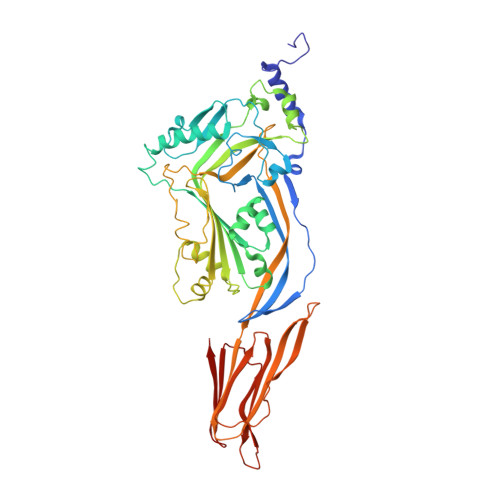 | |
UniProt | |||||
Find proteins for Q2XU26 (Streptococcus pneumoniae) Explore Q2XU26 Go to UniProtKB: Q2XU26 | |||||
Entity Groups | |||||
| Sequence Clusters | 30% Identity50% Identity70% Identity90% Identity95% Identity100% Identity | ||||
| UniProt Group | Q2XU26 | ||||
Sequence AnnotationsExpand | |||||
| |||||
| Length ( Å ) | Angle ( ˚ ) |
|---|---|
| a = 24.722 | α = 90 |
| b = 159.931 | β = 90 |
| c = 209.358 | γ = 90 |
| Software Name | Purpose |
|---|---|
| ADSC | data collection |
| MOLREP | phasing |
| PHENIX | refinement |
| HKL-2000 | data reduction |
| HKL-2000 | data scaling |